 Página anterior Página anterior |   Voltar ao início do trabalho Voltar ao início do trabalho | Página seguinte  |
A cultura do pêssego (Prunus persica (L.) Batsch var. persica) obteve grande impulso no Estado de São Paulo a partir da década de 60, com cultivares para inverno ameno do Planalto, tais como: 'Talismã', 'Maravilha', 'Joia', 'Ouromel' e 'Dourado', seguidas recentemente por outros tipos mais precoces e atraentes. As principais seleções de pêssego e nectarina adaptados a climas mais quentes foram obtidas no Instituto Agronômico (IAC), em seu programa de melhoramento genético iniciado em 1947 (Barbosa et al. 1997). Nos Estados Unidos, as cultivares de pêssego divergem geneticamente das cultivares Européias, em função de cruzamentos e seleção realizados (Arulsekar et al., 1986). Como a maioria dos paternais de pêssego do programa de melhoramento do Instituto Agronômico são de origem norte-americana, pode-se dizer que os genótipos brasileiros possuem base genética ampla. De acordo com Warburton e Bliss (1996), três dos 12 clusters obtidos com RAPD em pêssego, concentrou cultivares norte e latino americanas, alguns europeus e de ilhas do Pacífico, os demais nove apresentaram materiais da China, Paquistão, Rússia e Japão, sendo os chineses os materiais de maior diversidade genética. A cultivar brasileira Baronesa está no cluster II, onde se encontra o material norte americano com maior diversidade genética, se comparado com o cluster I.
O uso do RAPD tem sido utilizado com eficiência para identificação de cultivares de macieiras (Malus domestica Borkh.) (Koller et al., 1993), amoras vermelhas (Rubus idaeus L.) (Graham et al., 1994), ameixeiras (Prunus spp) (Ortiz et al., 1997) e roseiras (Rosa' hibrida) (Torres et al., 1993), sendo também um método rápido em relação ao RFLP e com maior número de marcadores em relação às isoenzimas. Pode ser muito útil na identificação de porta-enxertos, que são difíceis de identificar morfologicamente após a enxertia (Lu et al., 1996).
Este trabalho teve por objetivo a caracterização molecular através de RAPD e determinar a distância genética e o perfil dos marcadores de 'Tropical' e 'Douradão', juntamente com seus parentais e cultivares testemunhas, utilizadas como material para determinar distâncias genéticas.
Para extração de DNA, coletaram-se no Banco Ativo de Germoplasma de frutas de caroço do IAC, mantido no Núcleo de Agronomia de Capão Bonito, folhas jovens dos pessegueiros Douradão, Tropical, Dourado-1, Tutu, Aurora-1, Talismã e Flordaprince e da nectarineira Rubro-sol. Foram utilizadas 100 mg de folhas frescas maceradas em 350 l de tampão de extração DNAzol da GIBCO, adicionando-se 300 l de clorofórmio, centrifugando-se após cinco minutos e precipitando-se o sobrenadante com etanol por cinco minutos. Após a centrifugação, o "pellet" foi tratado com RNAse a 10mg/ml por 60 minutos para eliminar o RNA dos ácidos nucléicos e novamente centrifugado. O DNA do sobrenadante foi precipitado com 60 ml de NaCl de concentração 5M e 600 l de etanol 95% e, após 30 minutos, centrifugado e o "pellet" dissolvido em 100 l de 0.1 TE.
Na amplificação utilizou-se 8 ng de DNA e 23 ml do coquetel de amplificação com uma unidade de Taq DNA polimerase, denaturando o DNA a 94º C por dois minutos, seguido de 35 ciclos de 94ºC por 1 minuto, 35º C por 1 minuto, 72ºC por 1 minuto e 30 segundos, incubando a seguir com 72º C por 6 minutos. O produto amplificado foi colocado em gel de agarose a 1,5 %, submetido a eletroforese a 80 V por 3 horas, sendo revelado com brometo de etídio 0,25 % por uma hora e fotografado sob luz UV com filme Polaroid e filtro laranja. Foram testados 48 primers e selecionados os 7 altamente polimórficos para o estudo, com 31 bandas polimórficas.
Os resultados foram analisados pelo programa NTSYS versão 1.70 (Rohlf e Slice, 1992), utilizando as distâncias genéticas de Jaccard e aplicando o dendrograma de similaridade genética pelo método do UPGMA.
Na Tabela 1 estão os marcadores RAPD utilizados para caracterizar as cultivares pesquisadas. No pêssego 'Tropical' houve confirmação da maioria das marcas das suas avós 'Tutu' e 'Rubro-sol', mas algumas delas podem ter sido adquiridas durante a polinização aberta da seleção IAC 371-2, seu parental feminino (Figura 1), ou numa recombinação genética naquela geração. O mesmo se observou em 'Douradão' com relação a 'Dourado-1' em freqüência mais alta. Os avós destas seleções, 'Maravilha' e 'Tutu', confirmaram os marcadores em 'Dourado-1', exceto em OPAE11-450.
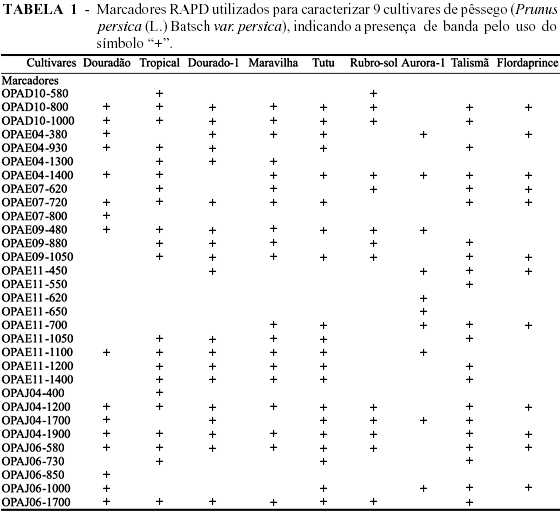
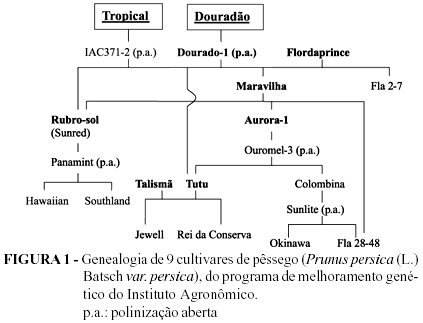
Excetuando 'Talismã', que é irmã de 'Tutu', as demais: 'Flordaprince', 'Rubro-sol' e 'Aurora-1', possuem um perfil de marcadores claramente distintos, evidenciados na Figura 2, onde estão mais destacadas no dendrograma de similaridade, mostrando uma maior distância das demais. O mesmo ocorreu com 'Douradão', com grande probabilidade de 'Dourado-1' ter sido polinizado por um material geneticamente distante. Evidencia-se na Figura 2 a similaridade de 'Tropical', 'Maravilha', 'Dourado-1', 'Tutu' e 'Talismã', cuja análise de genealogia confirma alto grau de parentesco, citado por Lu et al. (1996), que também encontraram relação entre marcadores RAPD e genealogia de 18 cultivares de pêssego porta enxerto, sendo que, os desvios observados no presente trabalho, se devem às polinizações abertas, tal como detectado por Warburton e Bliss (1996). A cultivar Aurora-1, também tem parentesco com 'Tutu' e a seleção Fla 28-48, que faz parte da genealogia do 'Douradão', é o cultivar com as maiores distâncias do grupo, talvez em função de polinizações abertas com material geneticamente distanciado.

As menores distâncias observadas (Tabela 2) foram entre 'Maravilha' e 'Tropical' (0,26), 'Maravilha' e 'Dourado-1' (0,27), e também entre 'Tutu' e 'Talismã', que são irmãos (0,29). Curiosamente, 'Tropical' não tem descendência de 'Maravilha', e sim de 'Rubro-sol', que por sua vez é parental de 'Maravilha'. Assim sendo, existe a possibilidade de 'Maravilha' ter participado da polinização aberta da seleção IAC371-2, mãe de 'Tropical', uma vez que 'Rubrosol' é geneticamente um pouco distante deste clone.
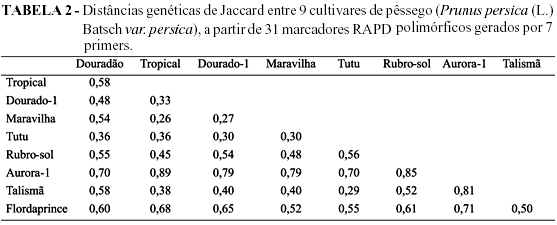
A análise de agrupamento não foi possível em função de um alto estresse e baixa correlação entre as distâncias reais e as calculadas. Neste trabalho se confirmou as observações de Ortiz et al. (1997), pela quais encontrou-se alto grau de polimorfismo no RAPD em 31 cultivares de ameixa e espécies relacionadas, a ponto de três primers serem suficientes para a identificação.
As conclusões deste trabalho são as seguintes:
- Os marcadores RAPD utilizados possibilitaram a indicação do grau de parentesco entre cultivares de pessegueiro;
- Mesmo entre as cultivares irmãs como 'Tutu' e 'Talismã' houve certa distância genética (0,29), mostrando que o método do RAPD pode ser usado para germoplasma aparentado;
- Os pêssegos 'Tropical' e 'Douradão' foram bem caracterizados em relação aos parentais e demais cultivares, o que permite utilizar a técnica para o registro das novas cultivares.
Léo Zimback (I); Wilson Barbosa (II,V); Edson Seizo Mori (III); Renato Ferraz de Arruda Veiga (IV)
wbarbosa[arroba]iac.sp.gov.br
I. Eng. Agr. Dr., Pesquisador Científico (PqC), Centro Experimental de Campinas (CEC) do Instituto Agronômico (IAC), telefone (19)32415188, Caixa Postal 28, CEP 13001-970, Campinas, SP. E-mail: lzimback[arroba]iac.br
II. Biólogo, M. Sc., PqC, CEC, IAC, telefone (19)32415188, E-mail: wbarbosa[arroba]iac.br
III. Eng. Florestal, Prof., Dr. da Faculdade de Ciências Agronômicas (FCA) da Universidade Estadual Paulista (UNESP), Botucatu, telefone (14)68027161, SP, Caixa Postal 237, CEP 18603-970, E-mail: esmori[arroba]fca.unesp.br
IV. Eng. Agr. Dr., PqC, CEC, IAC, telefone (19)32415188, E-mail: veiga[arroba]barao.iac.br
V. Bolsista do CNPq
 Página anterior Página anterior |   Voltar ao início do trabalho Voltar ao início do trabalho | Página seguinte  |
|
|
|